

发布时间:2019-06-10
6月10日,《自然》期刊在线发表了题为《DNA单碱基编辑技术引起RNA脱靶及其通过突变消除RNA活性》的研究论文,该研究由中国科学院脑科学与智能技术卓越创新中心(神经科学研究所)、上海脑科学与类脑研究中心、神经科学国家重点实验室、中国科学院灵长类神经生物学重点实验室杨辉研究组、四川大学华西二院/生命科学学院郭帆研究组和中国科学院上海营养与健康研究所隶属的计算生物学研究所(中国科学院-马普学会计算生物学伙伴研究所)李亦学研究组合作完成,该研究通过全转录组RNA测序发现DNA编辑工具单碱基编辑技术存在大量的RNA脱靶,首次证明了BE3、BE3-hA3A和ABE7.10等多个单碱基编辑技术均存在大量的RNA脱靶,并且ABE7.10还会导致大量的癌基因和抑癌基因突变,具有较强的致癌风险。该研究进而通过点突变的方式对三种单碱基编辑工具进行突变优化,使其完全消除RNA脱靶的活性,首次获得三种更高精度的单碱基编辑工具,为单碱基编辑基础进入临床治疗提供了重要的基础。
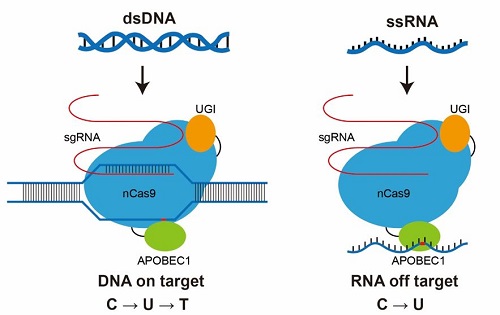
单碱基编辑技术是一种是自2012年CRISPR/Cas9技术被发现以来最寄予厚望的高精度基因编辑技术,其中一种单碱基编辑技术BE3可以在不切断DNA双链的情况精确的引入由C/G到T/A的点突变,另外一项单碱基编辑技术ABE7.10可以由T/A突变成C/G的技术,对于基因突变导致的遗传疾病的治疗具有重大意义。90%的罕见病无药可治,而单碱基编辑技术能够实现非常高精度的目标打靶,因此单碱基编辑技术相继成为脊髓性肌营养不良、地中海贫血、血友病、视网膜黄斑变性、遗传性耳聋等罕见病基因治疗的热门工具之一。
杨辉团队今年3月在《科学》杂志报道单碱基编辑技术BE3存在全基因组范围内的脱靶,引起了广泛的关注,已有的研究对于基因编辑工具的脱靶检测都瞄准在DNA水平。而杨辉团队这次将DNA编辑工具脱靶的检测范围扩展到RNA水平,首次证明常用的三种单碱基编辑技术均存在大量的RNA脱靶,通过精巧的实验设计证明了RNA脱靶主要是由于融合在Cas9上的脱氨酶导致。并且发现被寄予厚望的ABE7.10存在大量的RNA脱靶,并高频率地发生在癌基因和抑癌基因上。
通过对混合细胞和单细胞水平的RNA突变位点进行分析,该团队发现RNA突变位点和目的靶向序列没有相关性,是由脱氨酶产生的随机脱靶位点。因此,此前已经被应用到多种疾病模型上成功纠正遗传突变的单碱基编辑工具存在无法预测的RNA脱靶,有较大的致癌风险。
为了获得更加精准的单碱基编辑工具,杨辉研究组团队对单碱基编辑的胞嘧啶脱氨酶和腺嘌呤脱氨酶分别进行了突变优化,最终获得能够完全消除RNA脱靶并且维持DNA编辑活性的高精度单碱基编辑工具。除此之外,杨辉团队开发的ABE(F148A)突变体还能够缩小编辑窗口,实现更加精准的DNA编辑。该技术在特异性和精确性上超越了ABE7.10,有望在未来成为一种更加安全、更加精准的基因编辑工具,应用于临床治疗中。
该项工作由中科院脑智中心博士研究生周昌阳,计算生物学所博士研究生孙怡迪,四川大学生命科学学院学生燕蕊,上海科技大学生命学院博士研究生刘亚京,中国农业科学院深圳农业基因组研究所研究员左二伟等科研人员,在中科院脑智中心杨辉研究员,四川大学郭帆教授,中科院脑智中心周海波博士,计算生物学所李亦学研究员的共同指导下完成,课题组其他成员积极参与,并得到脑智中心流式分选平台,巴斯德所流式分选平台的大力协助,是众多课题组同理合作的重要成果。本工作得到国家高科技研发项目,中科院战略性先导科技专项,国家自然科学基金委员会,上海市科技重大项目,上海市科学技术委员会项目等项目的资助。
 附件下载:
附件下载: 