发布时间:2020-08-16
CRISPR/Cas9基因编辑技术以其高效性和特异性,自从2012年被发明以来备关注。学术界普遍认为基于CRISPR/Cas9及其衍生工具的临床技术将为人类的健康做出巨大贡献。然而值得注意的是,其脱靶风险一直令人担忧。将CRISPR/Cas9及其衍生工具用于临床,脱靶效应可能会引起包括癌症在内的很多副作用。研究人员推出过多种脱靶检测方法,然而这些方法都存在一些局限性,不能高灵敏性检测到脱靶突变,尤其是单核苷酸突变。因此关于CRISPR/Cas9及其衍生工具的真实脱靶率一直存在争议。精确,灵敏的脱靶检测技术是CRISPR/Cas9及其衍生工具应用于临床,治疗疾病的关键因素。
为了实现以上目标,科研人员建立了一种被命名为“GOTI”的新一代基因编辑脱靶检测技术。该技术使用携带CAG-LSL- tdTomato基因结构的Ai9转基因公鼠与野生型母鼠杂交产生具有CAG-LSL-tdTomato基因结构的受精卵。受精卵发育到2-cell后期,向一个卵裂球中注射含有Cre mRNA和基因编辑系统的RNA,Cre酶会剪切掉LSL结构, tdTomato基因开始表达红色荧光蛋白,构成一个基因编辑的细胞会被标记成红色的系统。胚胎在受体小鼠子宫内发育到胚胎14.5天,取出并将胚胎酶解消化成单细胞,流式细胞仪分选出红色荧光蛋白阳性和阴性细胞,分别抽取DNA,高通量全基因组测序。由于实验组和对照组来自同一枚受精卵,理论上基因背景完全一致,因此直接比对两组细胞的基因组,之间的差异可以认为是基因编辑工具造成的。
利用GOTI技术,研究人员发现经典的单碱基编辑工具BE3会导致基因组范围内完全随机无法预测的脱靶效应,不能应用于临床治疗 [1]。随后,利用高灵敏GOTI技术,在结构生物学指导下建立了新一代高保真单碱基编辑工具YE1-BE3-FNLS,解决了单碱基编辑工具的脱靶问题,打破了临床应用的瓶颈[2]。在GOTI技术支持下,建立A3G-CBE单碱基编辑系统,解决CCC motif编辑特异性问题[3]。相关研究成果分别发表于Science、Nature Methods和Science Advance杂志。
2020年8月14日,应Nature Protocols期刊邀请,中国科学院脑科学与智能技术卓越创新中心(神经科学研究所)、上海脑科学与类脑研究中心杨辉研究组、中国科学院上海营养与健康研究所隶属的计算生物学研究所(中国科学院-马普学会计算生物学研究所)李亦学研究组、斯坦福大学Lars M. Steinmetz研究组合作以“GOTI, a method to identify genome-wide off-target effects of genome editing in mouse embryos”为题,详细描述GOTI技术细节,与全球基因编辑领域科学家共享高精度,高灵敏性脱靶效应检测技术。
该研究杨辉研究员,李亦学研究员,Lars M. Steinmetz教授为共同通讯作者,左二伟研究员,孙怡迪研究员,魏武研究员,袁堂龙助理研究员为共同第一作者。本项目得到科技部,中科院,上海市的资助。
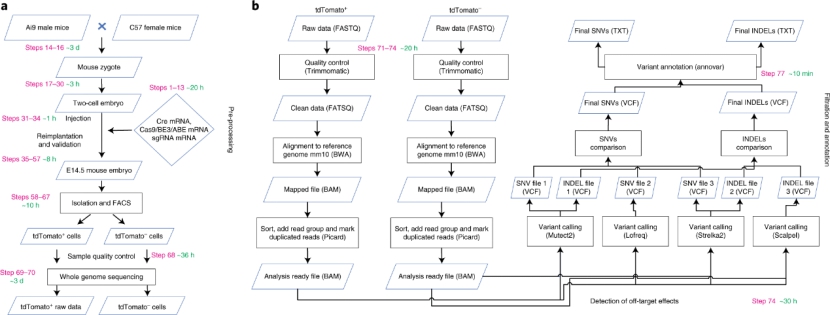
GOTI技术细节:a.小鼠2-cell注射,高通量测序;b.生信分析。
参考文献:
1. Zuo, E., Sun, Y., Wei, W., Yuan, T., Ying, W., Sun, H., Yuan, L., Steinmetz, L. M., Li, Y. & Yang, H. (2019) Cytosine base editor generates substantial off-target single-nucleotide variants in mouse embryos. Science. 364:289-2922.
2. Zuo, E., Sun, Y., Yuan, T., He, B., Zhou, C., Ying, W., Liu, J., Wei, W., Zeng, R., Li, Y. & Yang, H. (2020) A rationally engineered cytosine base editor retains high on-target activity while reducing both DNA and RNA off-target effects. Nature Methods. 17:600–604
3. Lee, S., Ding, N., Sun, Y., Yuan, T., Li, J., Yuan, Q., Liu, L., Yang, J., Wang, Q., Kolomeisky, A. B., Hilton, I. B., Zuo, E. & Gao, X. (2020) Single C-to-T substitution using engineered APOBEC3G-nCas9 base editors with minimum genome- and transcriptome-wide off-target effects. Science Advances. 6:eaba1773
 附件下载:
附件下载: